Pour contrer SARS-CoV-2, des simulations dévoilent comment une de ses protéines s’arrime à notre ARN
Comme tous les virus, le SARS-CoV-2 produit des protéines puis les utilise pour reconnaître et infecter nos cellules. Un mécanisme que certains médicaments ciblent et empêchent, mais encore mal connu dans le cas du nouveau coronavirus, apparu depuis moins d’un an. Grâce à des simulations moléculaires sur le supercalculateur Jean Zay, des chercheurs du LPCT (CNRS/Université de Lorraine), du CRAN (CNRS/Université de Lorraine), de l’université d’Alcalá (Espagne) et de l’université de Palerme (Italie) ont identifié deux modes d’accrochage d’une protéine virale, appelée SARS Unique Domaine, sur notre ARN. Selon ces travaux publiés dans le Journal of Physical Chemistry Letters, cette information aiderait à mieux cibler la recherche de médicaments contre le COVID-19.
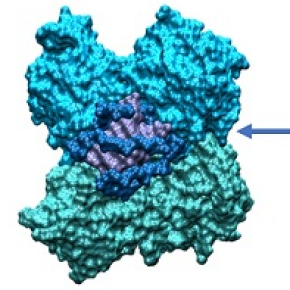
Responsable de la pandémie de COVID-19, le virus Sars-CoV-2 doit identifier les cellules qu’il souhaite infecter pour s’y reproduire. Pour cela, il fabrique des protéines qui, comme par tâtonnement, essayent de reconnaître chimiquement les cellules en s’imbriquant sur leurs récepteurs. Alors qu’une première protéine, appelée « spike », est chargée de reconnaître les cellules humaines, la protéine SARS Unique Domain permet au virus de se cacher de notre système immunitaire. Pour se camoufler, elle se replie sur elle-même en se fixant à l’ARN humain, ce qui empêche les lymphocytes de s’y accrocher et d’ainsi repérer le virus situé à côté. Afin de mieux comprendre ce mécanisme et de le déjouer, des chercheurs du Laboratoire de physique et chimie théoriques (LPCT, CNRS/Université de Lorraine), du Centre de recherche en automatique de Nancy (CRAN, CNRS/Université de Lorraine), de l’université d’Alcalá (Espagne) et de l’université de Palerme (Italie) ont mené des simulations moléculaires qui ont montré comment SARS Unique Domain s’arrime à l’ARN, une information cruciale pour espérer bloquer ce mode d’infection du virus.
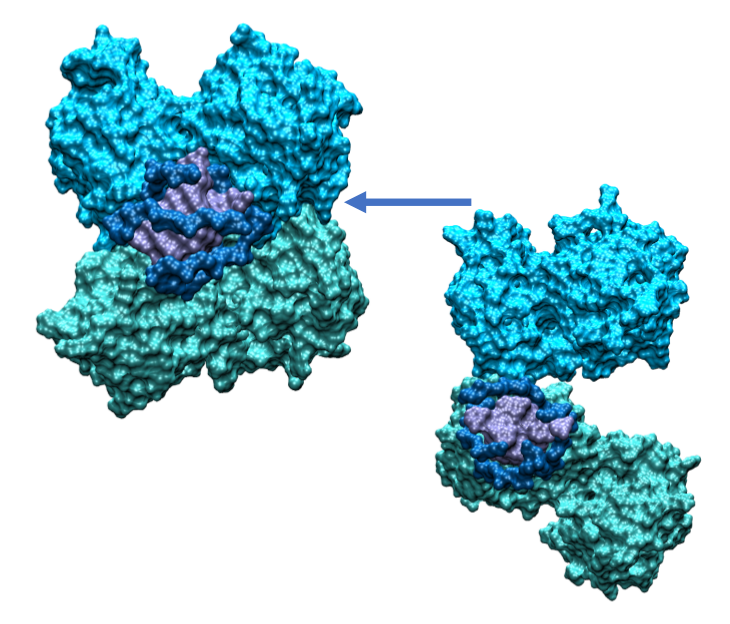
L’équipe, réunie sous le nom de projet SeekAndDestroy, a pour cela effectué des simulations extrêmement détaillées sur le supercalculateur Jean Zay, un équipement de pointe mis à disposition par le CNRS pour plusieurs équipes scientifiques luttant contre le COVID-19. Ce travail a montré que SARS Unique Domain pouvait s’accrocher aux quadruplexes de guanine de l’ARN, des arrangements en forme carrée des acides nucléiques. SARS Unique Domaine s’y attache de deux manières : le mode monomère, où une seule extrémité de la protéine est accrochée, et le mode dimère où ses deux extrémités sont arrimées au quadruplexe. Les chercheurs pensent que le mode monomère est employé en premier pour reconnaître et recruter l’ARN, puis le mode dimère permet à SARS Unique Domaine de se replier afin de rester incognito vis-à-vis du système immunitaire. L’identification de ce mode d’action offre une nouvelle piste pour lutter contre COVID-19. Les chercheurs ont utilisé la simulation moléculaire pour étudier aussi les modes d’action d’autres protéines du coronavirus, dont « spike », et ont aussi commencé à tester des molécules bloquant l’accrochage de SARS-CoV-2 aux cellules humaines.
Référence
Cécilia Hognon, Tom Miclot, Cristina Garcı́a-Iriepa, Antonio Francés-Monerris, Stephanie Grandemange, Alessio Terenzi, Marco Marazzi, Giampaolo Barone, and Antonio Monari. Role of RNA Guanine Quadruplexes in Favoring the Dimerization of SARS Unique Domain in Coronaviruses. J. Phys. Chem. Lett. 2020, 11, XXX, 5661–5667