Un laboratoire alsacien parmi les vainqueurs d'un défi science ouverte pour la conception de candidats-médicaments contre la maladie de Parkinson
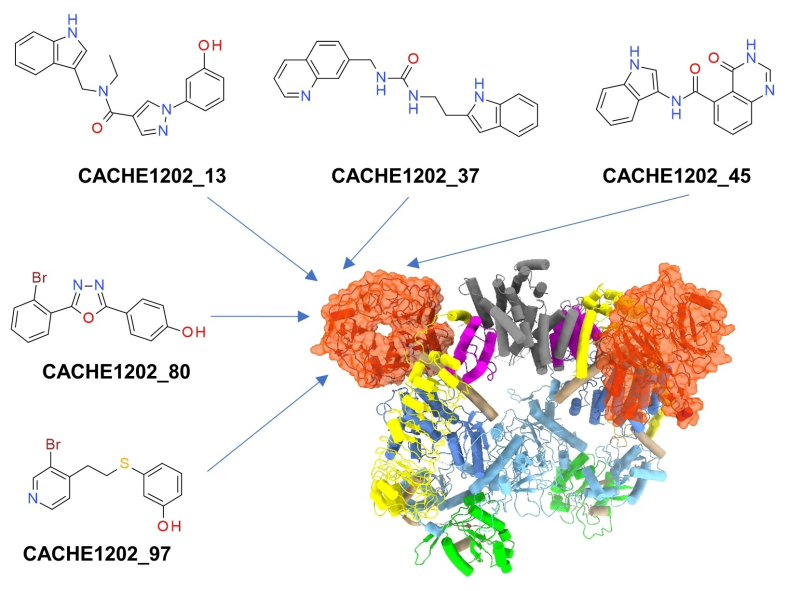
Dans le cadre d'une compétition internationale, des chercheurs du Laboratoire d’innovation thérapeutique (CNRS/Université de Strasbourg) ont combiné la modélisation et l’intelligence artificielle pour concevoir 5 nouvelles molécules qui se lient spécifiquement à une protéine cible impliquée dans la maladie de Parkinson.
Le challenge international CACHE #1, géré par le Structural Genomics Consortium et financé par la Fondation Michael J. Fox, est un défi scientifique ouvert auquel ont participé 23 laboratoires publics et privés. L’objectif : utiliser des méthodes de modélisation et d'intelligence artificielle pour découvrir des candidat-médicaments pour traiter une forme héréditaire de la maladie de Parkinson. Chaque participant a pu proposer jusqu'à 150 molécules qui ont été synthétisées puis testées pour leur aptitude à se lier à une protéine cible (LRKK2) fréquemment mutée dans une forme familiale de la maladie de Parkinson. Après 2 ans de validations expérimentales, un jury international de 5 experts de l'industrie pharmaceutique a pu classer les contributions de chacun en fonction des résultats obtenus (ex: nouveauté chimique, puissance de l'effet, sélectivité, etc…). Parmi les 6 laboratoires déclarés vainqueurs (5 publics, 1 privé) figure le Laboratoire d'innovation thérapeutique (LIT, CNRS/Université de Strasbourg) situé à la faculté de Pharmacie d'Illkirch. L’équipe du LIT a été sélectionnée pour son utilisation d'une méthode originale d'intelligence artificielle qui a conduit à la découverte de 5 candidats prometteurs.
Pour en savoir plus