IA et candidats-médicaments : une nouvelle méthode de conception par traitement numérique d'images
En alliant vision par ordinateur et intelligence artificielle (IA), des chimistes du LIT (CNRS/Université de Strasbourg) ont réussi à concevoir des nouvelles molécules-médicaments spécialement adaptées à leur cible en assimilant celles-ci à de simples images tridimensionnelles idéalisées. Des inhibiteurs puissants d’une protéine d’intérêt pharmaceutique impliquée dans la maladie orpheline de Blackfan-Diamond ont ainsi pu être conçus par ordinateur puis synthétisés et testés avec un minimum de ressources en un temps record. Ces résultats sont parus dans le Journal of Medicinal Chemistry.
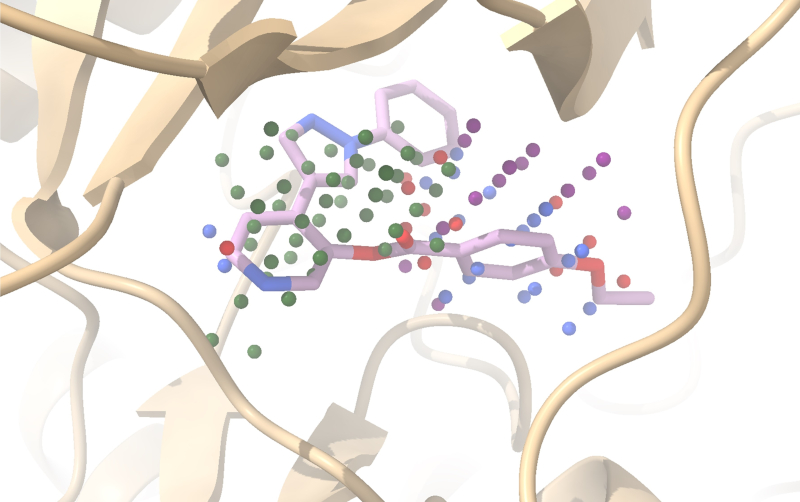
Lorsqu’il s’agit de concevoir de nouvelles molécules comme candidats-médicaments, tout l’enjeu consiste à correctement en prédire l'activité médicamenteuse, c’est-à-dire la possibilité pour une petite molécule de reconnaitre de manière spécifique une cible (protéine, ARN) et s’y insérer un peu à la façon d’une clef dans une serrure. Le degré avec lequel la petite molécule se lie au site de liaison cible, qui est une cavité ou une poche faisant partie d'une macromolécule beaucoup plus grande, définit la qualité de la molécule-médicament. Jusqu'à présent, la totalité des informations structurales connues sur la manière dont un médicament s'accroche à sa cible n'était utilisée que de manière parcellaire dans la conception de nouveaux candidats.
En assimilant une cible à une simple image tridimensionnelle, des chercheurs du Laboratoire d’innovation thérapeutiques (CNRS/Université de Strasbourg) ont réussi à inventorier sous forme d'un album photographique plus de 5 000 protéines cibles connues. En associant ces images de protéines à la structure des molécules s'y fixant, une méthode de vision par ordinateur a permis de développer des clichés représentant des petites fractions de molécules (appelées fragments) liées à des parties d'images (appelées sous-poche protéique). On peut ensuite assembler ces images pièce par pièce afin de proposer une molécule idéalement complémentaire de la protéine que l'on veut activer ou inhiber.
En se basant sur le principe de similarité ("des molécules similaires se lient à des protéines similaires"), ce jeu de LEGO® moléculaire, qui a en mémoire toutes les situations protéine-ligand connues, peut en effet proposer les molécules les plus adaptées à la protéine d'intérêt (image idéalisée) en plaçant au meilleur endroit possible les meilleurs fragments moléculaires que l’on peut d'assembler par intelligence artificielle. Cette nouvelle méthode de vision par ordinateur a ensuite été appliquée à la protéine cible CDK8 impliquée dans la maladie orpheline de Blackfan-Diamond, qui s’accompagne d’une incapacité pour la moelle osseuse de produire des globules rouges. En un temps record, pas moins de 43 molécules ont été produites dont plusieurs se sont avérées être de puissants inhibiteurs de CDK8. La méthode est applicable à toute cible dont la structure tridimensionnelle est connue et n'a aucun préjugé quant au type de molécules à imaginer. Elle est notamment capable d'explorer un espace chimique de plusieurs milliards de molécules possibles et permet donc de réduire considérablement le temps nécessaire à la synthèse et l'étude des toutes premières molécules candidates dont il faudra par la suite optimiser les propriétés afin d'en faire un réel candidat-médicament.
Référence
Target-focused library design by pocket-applied computer vision and fragment deep generative linking
Merveille Eguida, Christel Schmitt-Valencia, Marcel Hibert, Pascal Villa and Didier Rognan, Journal of Medicinal Chemistry octobre 2022.
https://doi.org/10.1021/acs.jmedchem.2c00931
