Cibler des repliements de l’ADN pour lutter contre le cancer
Face au fléau qu’est le cancer, la recherche redouble d’efforts pour développer des thérapies novatrices et ciblées. Dans une étude publiée dans la revue Angewandte Chemie International Edition, une équipe internationale pilotée par des chimistes et biologistes du CNRS présente une nouvelle cible thérapeutique prometteuse sur l’ADN de cellules cancéreuses, mais aussi des molécules capables de s'y attaquer de manière sélective.
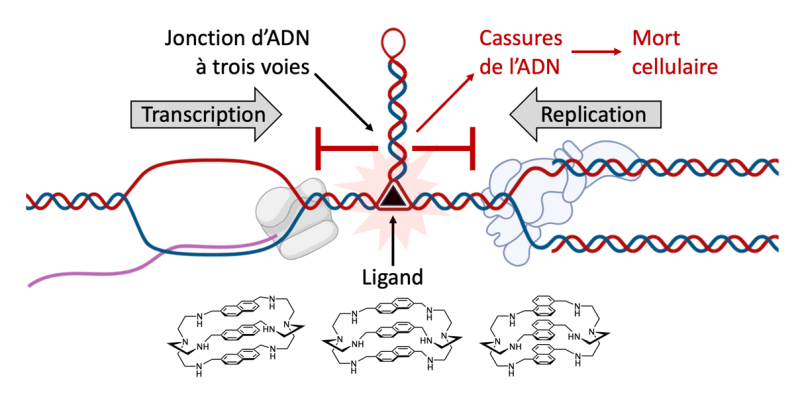
Les deux brins d’ADN qui s’assemblent en s’enroulant l’un autour de l’autre pour former la double hélice (ou duplexe) d’ADN sont souvent considérés comme inséparables. Ils sont pourtant régulièrement séparés, notamment lors de la réplication de l’ADN, mais sont alors placés sous étroite surveillance. Pourquoi ? Parce que, lors de cette séparation, les briques élémentaires qui composent un brin d’ADN, les « nucléobases », ne sont plus associées à leur partenaire privilégié sur le brin complémentaire (l’adénine avec la thymine, la guanine avec la cytosine) ; elles peuvent alors chercher à s’associer avec des nucléobases présentes sur le même brin, ce qui conduit à la formation de ‘repliements’ atypiques du brin d’ADN. La nature et la géométrie de ces repliements, ou « structures alternatives » du fait qu’elles diffèrent du duplexe, sont dictées par la nature même des séquences d’ADN impliquées.
Ces structures atypiques sont en soi problématiques car elles représentent un obstacle au passage (et donc au fonctionnement) des polymérases, les enzymes en charge des grandes manœuvres génomiques que sont la réplication, la transcription et la réparation de l’ADN. Mais vu sous un angle thérapeutique, elles pourraient bien être une aubaine. Si elles persistent, elles peuvent en effet entraîner des dommages à l’ADN et créer une instabilité génétique qui peut mettre en péril la survie de la cellule. Alors pourquoi ne pas justement chercher à stabiliser de telles structures sur l’ADN de cellules cancéreuses, par exemple ? Les cellules tumorales sont d’ailleurs particulièrement bien placées pour répondre à cette stratégie. D’abord parce que leur prolifération sollicite davantage les grandes manœuvres génomiques (la réplication en particulier). Mais aussi parce qu’elles présentent une amplification des régions capables de former des repliements alternatifs, ainsi que des mutations qui perturbent les mécanismes de réparation de l’ADN. Une stratégie anticancéreuse innovante consisterait donc à stabiliser certaines structures alternatives de l’ADN à l’aide de petites molécules pour créer des dommages qui pourront être réparés ailleurs, mais pas dans les cellules tumorales. C’est précisément l’étude menée par une équipe interdisciplinaire et internationale de chimistes et biologistes.*
Les scientifiques ont pour cela ciblé un type de repliement particulier : une structure en forme d’étoile à trois branches appelée jonction d’ADN à trois voies. Cette structure est intéressante car elle possède en son centre une cavité qui a la forme d’un prisme, ce qui ouvre la voie à un ciblage spécifique par des petites molécules (ou ligands)... également en forme de prismes ! Des spécialistes de la synthèse de ce type de ligands prismatiques ont préparé une série d’azacryptands.** Leurs interactions avec l’ADN ont été évaluées in vitro pour vérifier la sélectivité vis-à-vis des jonctions d’ADN par rapport aux autres structures présentes dans la cellule, en particulier le duplexe d’ADN. Les scientifiques ont ensuite démontré que ces molécules, en stabilisant les jonctions à trois voies, inhibent efficacement l’action des enzymes en charge des manœuvres génomiques. Les mécanismes d’interactions ligand/jonction ont pu être élucidés grâce à des calculs théoriques combinés à des résultats expérimentaux obtenus par résonance magnétique nucléaire (RMN). Enfin, les propriétés antiprolifératives des ligands les plus prometteurs ont été étudiées, ce qui a permis de mettre en évidence une toxicité plus importante vis-à-vis des cellules tumorales que des cellules saines.
Cette étude parue dans la revue Angewandte Chemie International Edition démontre l’intérêt des jonctions d’ADN à trois voies comme nouvelles cibles en oncologie. Elle propose également des agents thérapeutiques potentiels au mode d’action tout à fait original qui les stabilisent. L’arsenal d’outils thérapeutiques ciblés contre le cancer n’a pas fini de s’agrandir.
*De l’Institut de chimie moléculaire de l’Université de Bourgogne (CNRS/Université de Bourgogne), du laboratoire Chimie et modélisation pour la biologie du cancer (CNRS/INSERM/Institut Curie/Université Paris Saclay), de l’Institut de pharmacologie et de biologie structurale (CNRS/Université de Toulouse Paul Sabatier), du laboratoire PASTEUR (CNRS/ENS-PSL/Sorbonne Université) et du Central European Institute of Technology (CEITEC) en République Tchèque
**Les azacryptands sont des molécules polycycliques dont la structure définit une cavité à même de complexer fermement des ions (une crypte), dont l’origine remonte aux cages moléculaires initialement développées par le Prof. Jean-Marie Lehn (Prix Nobel de Chimie en 1987).
Rédacteur : AVR
Référence
Structural Optimization of Azacryptands for Targeting Three-Way DNA Junctions
Angélique Pipier, Titouan Chetot, Apollonia Kalamatianou, Nicolas Martin, Maëlle Caroff, Sébastien Britton, Nicolas Chéron, Lukáš Trantírek, Anton Granzhan & David Monchaud
Angew. Chem. Int. Ed. 2024
https://doi.org/10.1002/anie.202409780